
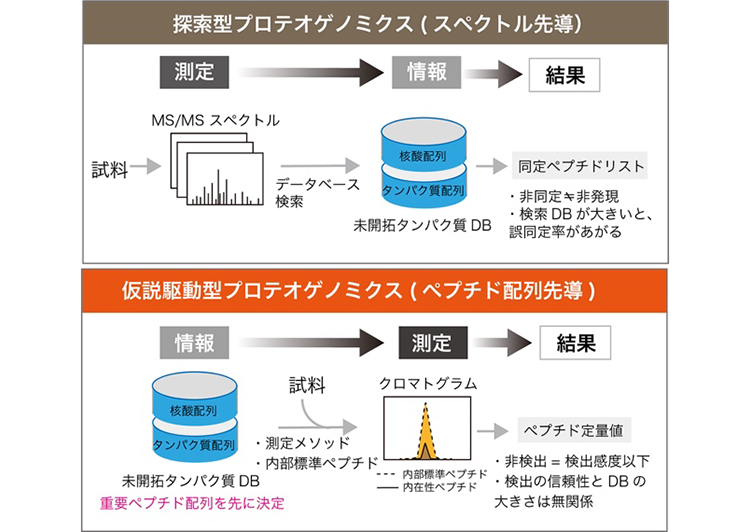
近年、未知のオープンリーディングフレームの発見や非典型的な翻訳開始や終結などが多数見出されており、実際のプロテオームはより複雑でこれまでの常識の範囲の中では探し出せないタンパク質を多数含んでいる可能性が高まっている。このような未知のプロテオームの探索には、核酸オミクス解析等で得られた情報を用いて質量分析データを解析する、いわゆるプロテオゲノミクスの利用が考えられるが、さまざまな技術的限界が存在する。本研究は、これらの既存のプロテオゲノミクスの技術的問題を克服するため、われわれが独自に構築した大規模絶対定量プロテオーム解析技術iMPAQT (in vitro proteome assisted MRM for Protein Absolute QuanTification) 法 【Matsumoto et al. Nat. Methods 2017】を発展させ、既存のプロテオゲノミクス研究と全く異なる思想に基づいたプロテオゲノミクスの枠組みである『仮説駆動型プロテオゲノミクス解析基盤』を創出する。構築された技術基盤を領域全体で共有し、未開拓プロテオームの全貌解明を目指す。
- Johmura Y, Yamanaka T, Omori S, Wang T, Sugiura Y, Matsumoto M, Suzuki N, Kumamoto S, Yamaguchi K, Hatakeyama S, Takami T, Yamaguchi R, Shimizu E, Ikeda K, Okahashi N, Mikawa R, Suematsu M, Arita M, Sugimoto M, Nakayama KI, Furukawa Y, Imoto S, *Nakanishi M. Senolysis by glutaminolysis inhibition ameliorates various age-associated disorders. Science 371(6526), 265-70 (2021)
- Kito Y, *Matsumoto M, Hatano A, Takami T, Oshikawa K, Matsumoto A, *Nakayama KI. Cell cycle-dependent localization of the proteasome to chromatin. Sci. Rep. 10(1), 5801 (2020)
- Oshikawa K, *Matsumoto M, Kodama M, Shimizu H, *Nakayama KI. A fail-safe system to prevent oncogenesis by senescence is targeted by SV40 small T antigen. Oncogene 39(10), 2170-86 (2020)
- *Matsumoto M, *Nakayama KI. The promise of targeted proteomics for quantitative network biology. Curr. Opin. Biotechnol. 54, 88-97 (2018)
- Morita M, Sato T, Nomura M, Sakamoto Y, Inoue Y, Tanaka R, Ito S, Kurosawa K, Yamaguchi K, Sugiura Y, Takizaki H, Yamashita Y, Katakura R, Sato I, Kawai M, Okada Y, Watanabe H, Kondoh G, Matsumoto S, Kishimoto A, Obata M, Matsumoto M, Fukuhara T, Motohashi H, Suematsu M, Komatsu M, Nakayama KI, Watanabe T, Soga T, Shima H, Maemondo M, *Tanuma N. PKM1 confers metabolic advantages and promotes cell-autonomous tumor cell growth. Cancer Cell 33, 355-67.e7 (2018)
- Kodama M, Oshikawa K, Shimizu H, Yoshioka S, Takahashi M, Izumi Y, Bamba T, Tateishi C, Tomonaga T, *Matsumoto M, *Nakayama KI. A shift in glutamine nitrogen metabolism contributes to the malignant progression of cancer. Nat. Commun. 11(1), 1320 (2020)
- Hata K, *Izumi Y, Hara T, *Matsumoto M, Bamba T. In-line sample processing system with an immobilized trypsin-packed fused-silica capillary tube for proteomic analysis of a small number of mammalian cells. Anal. Chem. 92(4), 2997-3005 (2020)
- Moriya Y, Kawano S, Okuda S, Watanabe Y, Matsumoto M, Takami T, Kobayashi D, Yamanouchi Y, Araki N, Yoshizawa AC, Tabata T, Iwasaki M, Sugiyama N, Tanaka S, *Goto S, *Ishihama Y. The jPOST environment: an integrated proteomics data repository and database. Nucleic Acids Res. 47(D1), D1218-24 (2018)
- Matsumoto M, Matsuzaki F, Oshikawa K, Goshima N, Mori M, Kawamura Y, Ogawa K, Fukuda E, Nakatsumi H, Natsume T, Fukui K, Horimoto K, Nagashima T, Funayama R, Nakayama K, *Nakayama KI. A large-scale targeted proteomics assay resource based on an in vitro human proteome. Nat. Methods 14, 251-8 (2017)
- Matsumoto A, †Pasut A, †Matsumoto M (†equally contributed), †Yamashita R, Fung J, Monteleone E, Saghatelian A, Nakayama KI, Clohessy JG, *Pandolfi PP. mTORC1 and muscle regeneration are regulated by the LINC00961-encoded SPAR polypeptide. Nature 541, 228-32 (2017)

