
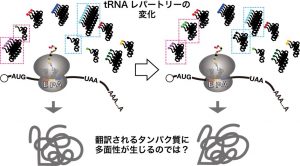
タンパク質のあるアミノ酸残基が特定の同義コドンによってコードされることは、タンパク質の翻訳速度や精度の違いを介してフォルディングや細胞内局在など、翻訳以降の様々な面へ影響する場合があることが指摘されている。事実、それまでタンパク質の機能には影響しないと思われていたsynonymous mutationには、様々な遺伝病に至る変異があることが明らかとなっている。今までコドン選択はそのコドンをデコードするisoacceptor tRNA(アンチコドン配列で規定されるtRNA種)の量に対応すると考えられており、各isoacceptor tRNA量の総体(tRNAレパートリー)はゲノム上のtRNA遺伝子の数で決定されるため生物種で一定だと考えられてきた。しかし、近年、tRNAレパートリーが内的外的環境によって変化することが明らかとなり、この変化を利用したproteome形成制御の可能性が示唆されている。
我々は長年、tRNAの様々な動態について解析してきたが、強固な高次構造と多様な修飾によって網羅的解析法では定量の難しかったtRNAの新規絶対定量法−OTTER法−を開発した。その中で、出芽酵母においてminor isoacceptor tRNAが呼吸条件下では軒並み発酵条件の2倍程度に増え、major isoacceptor tRNAはこれを相殺するように減少することを見出した(Nagai et al., RNA, 2021)。これによってtRNA環境適合性指標であるtAIが呼吸条件下で上昇するトップ100 ORFを調べると、オルガネラタンパク質や天然変性領域を持つタンパク質等が偏って含まれることが判った。こうした翻訳延長環境の変化は、タンパク質の量的調節だけではなく、局在化や機能化等の質的な違い、すなわち、タンパク質にマルチファセット性を与える可能性がある。生物は、翻訳環境の違いによって生ずるタンパク質のマルチファセット性を積極的に利用しているのではないだろうか。こうした仮説を検証する目的で、本研究では出芽酵母を材料に下記の4項目の研究課題を進める。
【1】発酵条件と呼吸条件の間でのmRNAの翻訳状態の違いをribosome profilingで検討する。特に、ERやミトコンドリア上で翻訳共役的に膜透過するタンパク質の翻訳状態に着目する。
【2】呼吸条件下でtAIが上昇したミトコンドリアに局在するタンパク質群を対象に、局在化効率や、翻訳共役型膜透過の割合等が、炭素源に左右されるかを検討する。
【3】呼吸条件下でtAIが上昇した天然変性領域を含むタンパク質、特に核膜孔複合体(NPC)構成タンパク質(Nup)のNPCへの組み込み等が炭素源でどう影響されるか検討する。
【4】呼吸培地中で発現量の上昇するminor tRNA群を発酵培地中並みに抑える、またはこの逆の状態を達成し、前2項で取り上げたタンパク質群の局在化や機能化への影響を検討する。
特に、Nupが左右する核−細胞質間輸送担体の細胞内動態を詳しく解析し、tRNAレパートリーの制御がタンパク質の機能的な多面性を生み出すか検証する。
- Nagai A, Mori K, Shiomi Y, *Yoshihisa T. OTTER, a new method quantifying absolute amounts of tRNAs. RNA 27, 628-640 (2021)
- Matsuki Y, Matsuo Y, Nakano Y, Iwasaki S, Yoko H, Udagawa T, Li S. Saeki Y, Yoshihisa T, Tanaka K, Ingolia NT, *Inada T. Ribosomal protein S7 ubiquitination during ER stress in yeast is associated with selective mRNA translation and stress outcome. Sci Rep 12-e19669 (2020)
- Hayashi S, Mori S, Suzuki T, Suzuki T, *Yoshihisa T. Impact of intron removal from tRNA genes on Saccharomyces cerevisiae. Nucleic Acids Res 47, 5936-5949 (2019)
- *Yoshihisa T. Nucleocytoplasmic shuttling of tRNAs and implication of the cytosolic Hsp70 system in tRNA import. Nucleus 6, 339-343 (2015)
- Tsuboi T, Yamazaki R, Nobuta R, Ikeuchi K, Makino S, Ohtaki A, Suzuki Y, Yoshihisa T, Trotta C, *Inada T. The tRNA splicing endonuclease complex cleaves the mitochondria-localized CBP1 mRNA. J Biol Chem 290, 16021-16030 (2015)
- Takano A, Kajita T, Mochizuki M, Endo T, *Yoshihisa T. Cytosolic Hsp70 and co-chaperones constitute a novel system for tRNA import into the nucleus. eLife 4, e04659 (2015)
- Song J, Tamura, Y, Yoshihisa T, *Endo T. A novel import route for an N-anchor mitochondrial outer membrane protein aided by the TIM23 complex. EMBO Rep 15,670-677 (2014)
- *Yoshihisa T. Handling tRNA introns, archaeal way and eukaryotic way. Front Genet 5, e00213 (2014)
- Mori S, Kajita T, Endo T, *Yoshihisa T. The intron of tRNA-TrpCCA is dispensable for growth and translation of Saccharomyces cerevisiae. RNA 17, 1760-1769 (2011)
- Takano A, Endo T, *Yoshihihsa T. tRNA actively shuttles between the nucleus and cytosol in yeast. Science 309, 140-142 (2005)

