
翻訳の伸長反応は、ペプチド鎖のフォールディングや細胞内小器官への配送などと共役しており、その異常は不良タンパク質の産生に直結します。細胞はこれを回避するために、異常な翻訳伸長を感知し、強制的に翻訳を終結させる品質管理機構(RQC: Ribosome-associated Quality Control)を備えています。細胞内では同一のmRNAを複数のリボソームが翻訳しているため、異常な翻訳停滞が生じると停滞したリボソームと後続のリボソームが衝突し“リボソームの交通渋滞”が形成されます。RQCのセンサータンパク質であるHel2はこれを異常な翻訳と認識し、非典型的な翻訳終結反応によって停滞したリボソームを除去します。これにより、リボソームの交通渋滞が解消され、翻訳が再開されます。
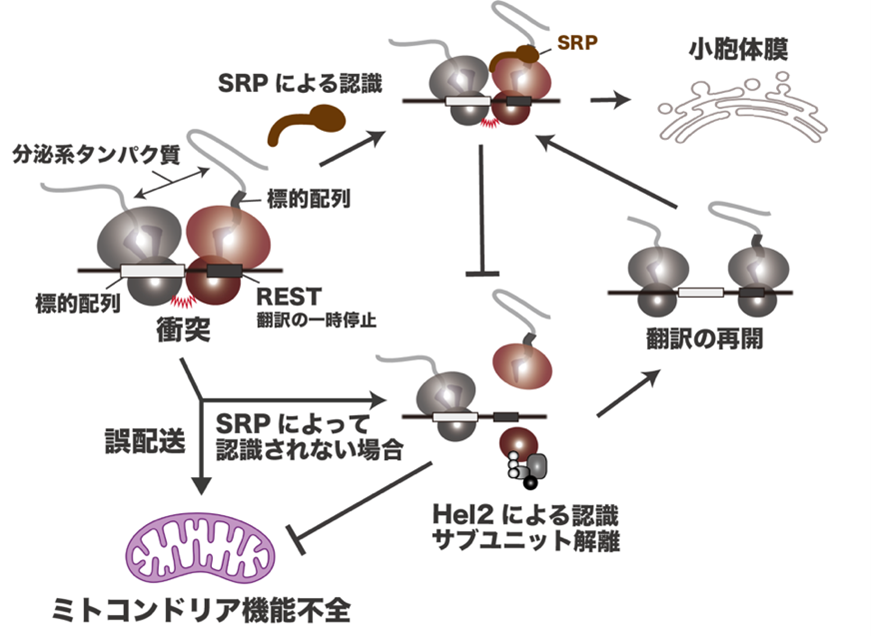
最近、わたしたちは、SRP (Signal Recognition Particle)を介した分泌系タンパク質の共翻訳的な配送異常がRQCに識別されることを見いだしました(Matsuo and Inada., Cell Rep. 2021)。テイルアンカー型を除く分泌系タンパク質の大部分は、SRPを介して共翻訳的に小胞体膜へと配送されます。その際、配送の補助機構として、分泌系タンパク質をコードするmRNAには翻訳の一時停止を誘導するレアコドンクラスター(REST)がコードされており、このRESTによってリボソームの交通渋滞が生じていることも報告されています。しかし、RQCによって識別される配送異常は、SRPによる認識に欠損があるものに限られており、小胞体膜への配送中にリボソームの交通渋滞が生じても、SRPの結合によってRQCの誘導がキャンセルされる可能性が示唆されています。そこで、本研究では、その分子機構の解明を目指し解析を進めています。また、SRPの識別に欠損があり、かつRQCによる除去が失敗した分泌系タンパク質は、ミトコンドリアへと誤配送されることがわかっています。本研究では、このミトコンドリアへと誤配送された分泌系タンパク質の運命についても解析を進めています。
- Best K, Ikeuchi K, Kater L, Best D, Musial J, Matsuo Y, Berninghausen O, Becker T, Inada T*, Beckmann R*. Structural basis for clearing of ribosome collisions by the RQT complex. Nat Commun. 14(1): 921. (2023)
- Tesina P#*, Ebine S#, Buschauer R#, Thoms M, Matsuo Y, Inada T*, Beckmann R*. Molecular basis of eIF5A-dependent CAT tailing in eukaryotic ribosome-associated quality control. Mol Cell. 83(4): 607-621. (2023)
- Matsuo Y*, Uchihashi T, Inada T*. Decoding of the ubiquitin code for clearance of colliding ribosomes by the RQT complex. Nat Commun. 14(1):79. (2023)
- Narita M#, Denk T#, Matsuo Y, Sugiyama T, Kikuguchi C, Ito S, Sato N, Suzuki T, Hashimoto S, Machova I, Tesina P, Beckmann R*, Inada T*. A distinct human disome collision interface harbors K63-linked polyubiquitination of uS10 to trigger hRQT-mediated subunit dissociation. Nat Commun. 13(1):6411. (2022)
- Li S#, Ikeuchi K#, Kato M, Buschauer R, Sugiyama T, Adachi S, Kusano H, Natsume T, Berninghausen O, Matsuo Y, Becker T, Beckmann R*, Inada T*. Sensing of individual stalled 80S ribosomes by Fap1 for non-functional rRNA turnover. Mol Cell. 82(18):3424-3437. (2022)
- Matsuo Y* and Inada* T. Ribosome collision sensor Hel2 recognizes mistargeting secretory ribosome-nascent chain complexes. Cell Rep. 34:108877. (2021)
- Buschauer R#, Matsuo Y# (#Co-first Author), Sugiyama T, Chen YH, Alhusaini N, Sweet T, Ikeuchi K, Cheng J, Matsuki Y, Nobuta R, Gilmozzi A, Berninghausen O, Tesina P, Becker T, Coller J*, Inada T*, Beckmann R*. The Ccr4-Not complex monitors the translating ribosome for codon optimality. Science. 368(6488):eaay6912 (2020)
- Matsuo Y#, Tesina P#, Nakajima S, Mizuno M, Endo A, Buschauer R, Cheng J, Shounai O, Ikeuchi K, Saeki Y, Becker T, Beckmann R*, Inada T*. RQT complex dissociates ribosomes collided on endogenous RQC substrate SDD1. Nat Struct Mol Biol. (4):323-332. (2020)
- Matsuo Y#, Ikeuchi K#, Saeki Y, Iwasaki S, Schmidt C, Udagawa T, Sato F, Tsuchiya H, Becker T, Tanaka K, Ingolia NT, Beckmann R, Inada T*. Ubiquitination of stalled ribosome triggers ribosome-associated quality control. Nat Commun. 8(1):159. (2017)
- Matsuo Y, Granneman S, Thoms M, Manikas RG, Tollervey D, Hurt E*. Coupled GTPase and remodelling ATPase activities form a checkpoint for ribosome export. Nature. 505(7481):112-116. (2014)
#Co-first author, *Corresponding author

