
ヒト細胞が作るタンパク質の約半数以上は複数のドメイン(タンパク質の構造の一部で他の部分とは独立した3次元構造をとる)からなるマルチドメインタンパク質である。試験管内の折り畳み実験ではこのようなタンパク質はドメイン間の相互作用によって凝集しやすい。しかし、細胞内ではこれらのタンパク質の多くは翻訳と共役して効率よく折り畳まれる。従って、その新生鎖上には、翻訳装置の活性を制御するなど、未同定の様々な仕組みが隠されていると予想される。
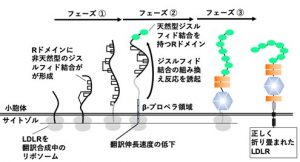
我々は、ヒト培養細胞の小胞体に送り込まれてくる翻訳途上のマルチドメインタンパク質LDL受容体(LDLR)にジスルフィド結合が導入され、正しい立体構造へと折り畳まれていく過程を、これまでにない詳細なレベルで可視化する系を構築した(Kadokura et al., PNAS 117, 16401-16408 (2020))。本研究では、この系を駆使するとともに、領域内での共同研究を進めて、翻訳と共役した、タンパク質の折り畳みを促進するために、新生鎖の配列中に隠された様々な仕組みを調べるとともに、その普遍性や病気との関連を解析し、誕生前のタンパク質のもつ新たな世界やその意義を明らかにしたい。
- *Kadokura H, Dazai Y, Fukuda Y, Hirai N, Nakamura O, Inaba K. Observing the nonvectorial yet cotranslational folding of a multidomain protein, LDL receptor, in the ER of mammalian cells. Proc Natl Acad Sci USA 117, 16401-16408 (2020)
- Fujimoto T, Inaba K, *Kadokura H. Methods to identify the substrates of thiol-disulfide oxidoreductases. Protein Sci. 28, 30-40 (2019) (Invited Review)
- Fujimoto T, Nakamura O, Saito M, Tsuru A, Matsumoto M, Kohno K, Inaba K, *Kadokura H. Identification of the physiological substrates of PDIp, a pancreas-specific protein disulfide isomerase family member. J. Biol. Chem. 293, 18421-18433 (2018)
- Tsuchiya Y, Saito M, Kadokura H, Miyazaki J-I, Tashiro F, Imagawa Y, Iwawaki T, *Kohno K. IRE1-XBP1 pathway regulates oxidative proinsulin folding in pancreatic β cells. J. Cell Biol. 217, 1287-1301 (2018)
- *Kadokura H, Saito M, Tsuru A, Hosoda A, Iwawaki T, Inaba K, *Kohno K. Identification of the redox partners of ERdj5 /JPDI, a PDI family member, from an animal tissue. Biochem. Biophys. Res. Commun. 440, 245-250 (2013)
- Chng SS, Xue M, Garner RA, Kadokura H, Boyd D, Beckwith J, *Kahne D. Disulfide rearrangement triggered by translocon assembly controls lipopolysaccharide export. Science 337, 1665-1668 (2012)
- Yanagitani K, Kimata Y, Kadokura H, *Kohno K. Translational pausing ensures membrane targeting and cytoplasmic splicing of XBP1u mRNA. Science 331, 586-589 (2011)
- *Kadokura H, *Beckwith J Mechanisms of oxidative protein folding in the bacterial cell envelope. Antioxid. Redox Signal. 13, 1231-1246 (2010)
- *Kadokura H, *Beckwith J. Detecting folding intermediates of a protein as it passes through the bacterial translocation channel. Cell 138, 1164-1173 (2009)
- Kadokura H, Tian H, Zander T, Bardwell JCA, *Beckwith J. Snapshots of DsbA in action: Detection of proteins in the process of oxidative folding. Science 303, 534-537 (2004)

